R pour les biostatistiques en 5 minutes chrono (!)
gilles.hunault "at" univ-angers.fr
Attention :
ceci est la suite de R15
donc lisez d'abord la page R15
Il y a une version anglaise disponible ici.
Table des matières cliquable
1. Pourquoi R pour les biostatistiques ?
2. Bibliographie restreinte et orientée biostatistiques
3. Démonstration via Datajoy
4. DémonstrationS via RStudio
1. Pourquoi R pour les biostatistiques ?
Les arguments cités en faveur de R pour la bioinformatique s'appliquent tout à fait à R pour les biostatistiques : R est gratuit et exhaustivement complet pour tout ce qui touche aux statistiques et aux biostatistiques. De plus toutes les méthodes modernes et récentes sont disponibles pour R, contrairement aux grands logiciels statistiques comme SAS, SPSS ou Statistica.
2. Bibliographie restreinte et orientée biostatistiques
Contrairement aux ouvrages dédiés à la bioinformatique avec R, il y a pléthore d'ouvrages dédiés aux biostatistiques avec R :

Des recherches sur le web avec les expressions
biostatistics, "with R", "using R" et books ou filetype:pdf aboutissent à des milliers d'ouvrages, parfois publiés, parfois simplement distribués sur le Web,
comme on peut le vérifier ci-dessous.
3. Démonstration via Datajoy
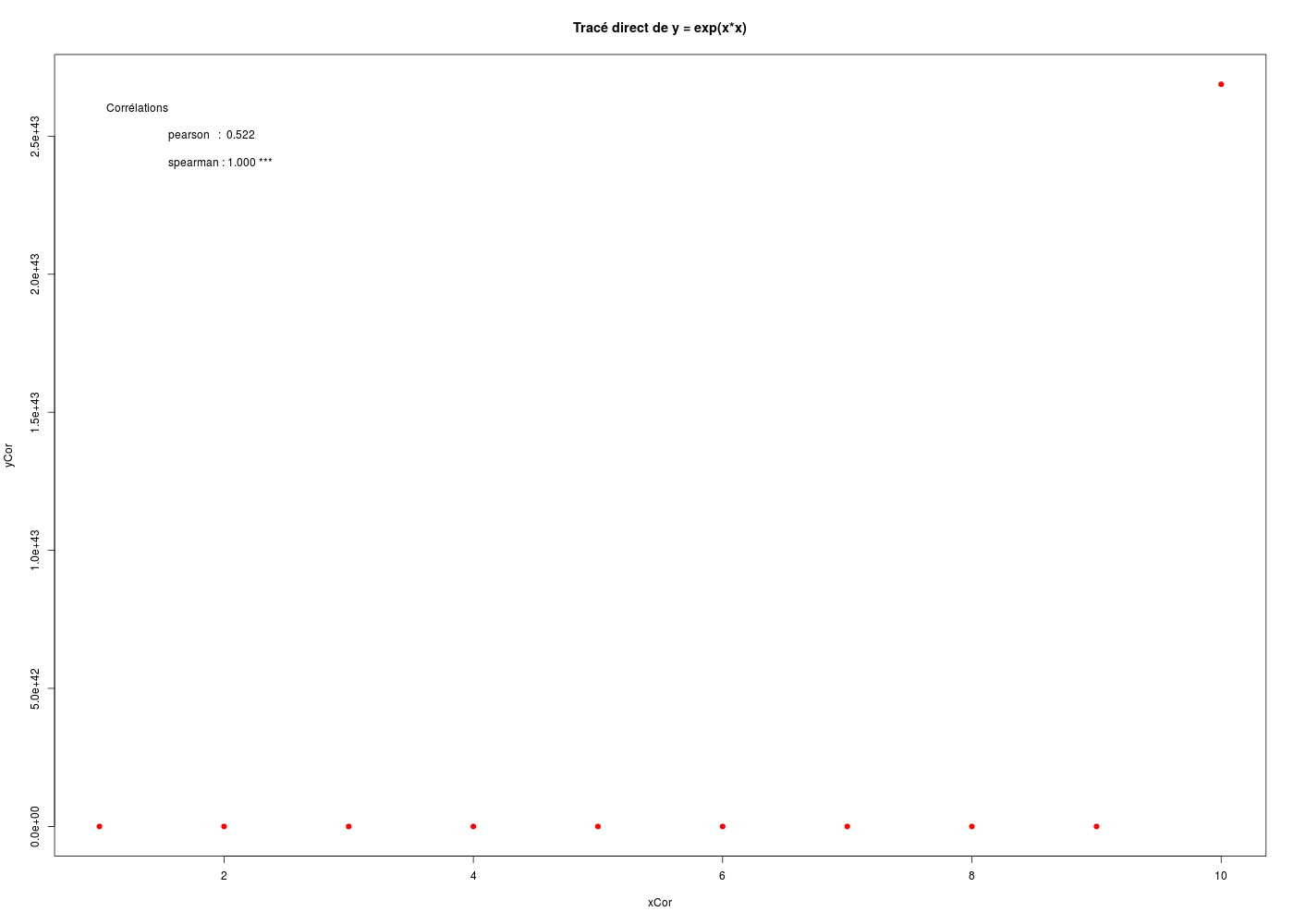
Voici un texte R simple à lire pour montrer la différence entre la corrélation au sens de Pearson, c'est-à-dire la corrélation linéaire et la corrélation de Spearman, c'est-à-dire la corrélation monotone des rangs (tout ce qui commence par # est un commentaire, ignoré par R) :
cat("Calculs de coeficients de corrélation\n")
# x et exp(x*x) sont liés mais pas linéairement
xCor <- 1:10
yCor <- exp(xCor*xCor)
# donc la corrélation au sens de Pearson est faible
corp <- cor(xCor,yCor,method="pearson")
corpf <- sprintf("%0.3f",corp)
pvcp <- cor.test(xCor,yCor,method="pearson")$p.value
cat(" pearson : ",corpf)
cat(" ; p-value = " ,sprintf("%0.3f",pvcp),"\n",sep="")
# alors que celle de Spearman est au maximum
cors <- cor(xCor,yCor,method="spearman")
corsf <- sprintf("%0.3f",cors)
pvcs <- cor.test(xCor,yCor,method="spearman")$p.value
cat(" spearman : ",corsf)
cat(" ; p-value = ",sprintf("%0.3f",pvcs),"\n",sep="")
cat("\n")
# vérifications par le tracé
titre <- "Tracé direct de y = exp(x*x)"
plot(xCor,yCor,main=titre,pch=19,col="red")
text(x=1.0,y=2.6*10**43,pos=4,adj=4,labels="Corrélations")
text(x=1.5,y=2.5*10**43,pos=4,adj=4,labels=paste("pearson : ",corpf))
text(x=1.5,y=2.4*10**43,pos=4,adj=4,labels=paste("spearman :",corsf,"***"))
# données utilisées
cat("Voici les valeurs de x et de y\n")
print(cbind(xCor,yCor),row.names=FALSE)
Voici le résultat de l'exécution :
Calculs de coeficients de corrélation
pearson : 0.522 ; p-value = 0.122
spearman : 1.000 ; p-value = 0.000
Voici les valeurs de x et de y
xCor yCor
[1,] 1 2.718282e+00
[2,] 2 5.459815e+01
[3,] 3 8.103084e+03
[4,] 4 8.886111e+06
[5,] 5 7.200490e+10
[6,] 6 4.311232e+15
[7,] 7 1.907347e+21
[8,] 8 6.235149e+27
[9,] 9 1.506097e+35
[10,] 10 2.688117e+43
Et la courbe cliquable (remarquer les valeurs sur l'axe Y) :
Un simple copier/coller du code R ci-dessus dans une fenêtre du site Datajoy permet de vérifier tout ceci sans aucune installation de R.
4. DémonstrationS via RStudio
Un point très fort et très important de R est sa réactivité. On trouve par exemple en R un package nommé survivalROC pour les AUROCS dépendantes du temps, ce que ni SAS, SPSS ou Statistica ne savent faire. Il suffit de regarder ici chaque jour quel package est mis à jour ou nouvellement mis à disposition pour se rendre compte que R évolue très vite, là où SAS, SPSS et Statistica ne fournissent qu'une mise à jour par an et encore, sans toujours beaucoup de nouveautés. De plus installer un nouveau package se fait en quelques secondes (au pire en quelques dizaines de secondes) avec Rstudio donc on dispose tout de suite de tout en R.
Un autre point très fort aujourd'hui (2016) est sa capacité à fournir de la recherche reproductible à moindre cout utilisateur. Ainsi le texte suivant qui est au format Markdown permet de produire, grâce aux deux instructions modifiables output: pdf_document et nbp <- 10, le document paramétré demor5Biostats.pdf.
Si on change les deux instructions en output: word_document et nbp <- 8, alors R produit le fichier Word suivant demor5Biostats.docx. Enfin, si on change ces deux instructions en output: html_document et nbp <- 20, alors R produit la page Web suivante demor5Biostats.html.
En moins de 10 secondes (le temps qu'il faut pour modifier output: et nbp et pour ré-éxecuter le code) R est donc capable de produire des documents externes différents avec le paramétrage souhaité.
Vous imaginez donc comme c'est facile d'automatiser un rapport des analyses de fichiers Excel pour un article sans copier/coller des résultats... Quel gain de temps ! Qui est contre de ne plus avoir à transférer des résultats d'un logiciel statistique dans son traitement de texte favori et d'employer son temps à des activités plus "intelligentes" ?
|
 Retour à la page principale de
(gH)
Retour à la page principale de
(gH)

